北京大学汪阳明、席建忠与清华大学张强锋团队合作揭开脊椎动物长非编码RNA保守性之谜
2024/01/24 信息来源: 未来技术学院
文字:张金博| 编辑:麦洛 | 责编:燕元人类基因组不仅编码蛋白质基因,还包含数以几十万计的长非编码RNA(lncRNA)基因,被称为生命“暗物质”。lncRNA的发现和表征是过去几十年分子生物学领域的重大进展。已有研究成果表明,lncRNA在发育、肿瘤等多种生理和疾病过程中发挥重要调控作用。分析基因在不同物种中的同源性和保守性,是研究和理解基因的基础功能和进化的重要手段。但是,与蛋白质基因相比,lncRNA的序列保守性极低,传统的序列比对方式只能鉴定出极少的不同物种间同源的lncRNA(图1)。例如,在斑马鱼和人类上万的lncRNA基因中,通过序列比对只能找到十几个序列保守的同源lncRNA。然而,已有的案例表明,即使是序列保守性很低的lncRNA,不同物种中的相关lncRNA仍然具有功能上的保守性。显然,lncRNA的保守性具有不同于蛋白质基因保守性的特征和要素,“这些特征和要素究竟是什么”是当前lncRNA领域亟需解开的一个谜团。

图1. 长非编码RNA保守性是领域之谜
1月9日,清华大学生命科学学院张强锋副教授,北京大学未来技术学院汪阳明教授、席建忠教授研究团队合作在Nature genetics上发表题为“Computational prediction and experimental validation identify functionally conserved lncRNAs from zebrafish to human”(计算预测和实验验证鉴定人类和斑马鱼之间功能保守的长非编码RNA)的研究论文。该工作开发了一套新的算法,在包括人类、小鼠、斑马鱼在内的8种脊椎动物中鉴定保守的同源lncRNA,同时开发了基于CRISPR的基因敲除和回补筛选系统,通过一系列实验验证了所鉴定的同源lncRNA在不同物种中的功能保守性,为该领域的研究提供了新的思路。
算法开发:该团队首先开发了一套鉴定不同物种之间同源lncRNA的计算方法(lncHOME)。lncHOME计算方法通过比较基因组和机器学习的人工智能方法,在8种脊椎动物中鉴定出了一类在不同物种中具有保守基因组位置及保守RNA结合蛋白(RBP)结合位点模式的lncRNA(图2)。这些不同物种中潜在同源的lncRNA被命名为coPARSE-lncRNA 【lncRNA with conserved genomic locations and patterns of RNA binding protein (RBP) binding sites】。lncHOME计算方法鉴定了570个在斑马鱼中具有同源基因的人类coPARSE-lncRNA,与之相比,通过传统的序列比对的方式仅能鉴定出17个序列保守的同源lncRNA。相比于非同源的lncRNA,这些coPARSE-lncRNA基因富集了更多疾病相关突变,更倾向于在癌症组织中异常表达,预示着coPARSE-lncRNA可能具有重要的生理或病理功能。
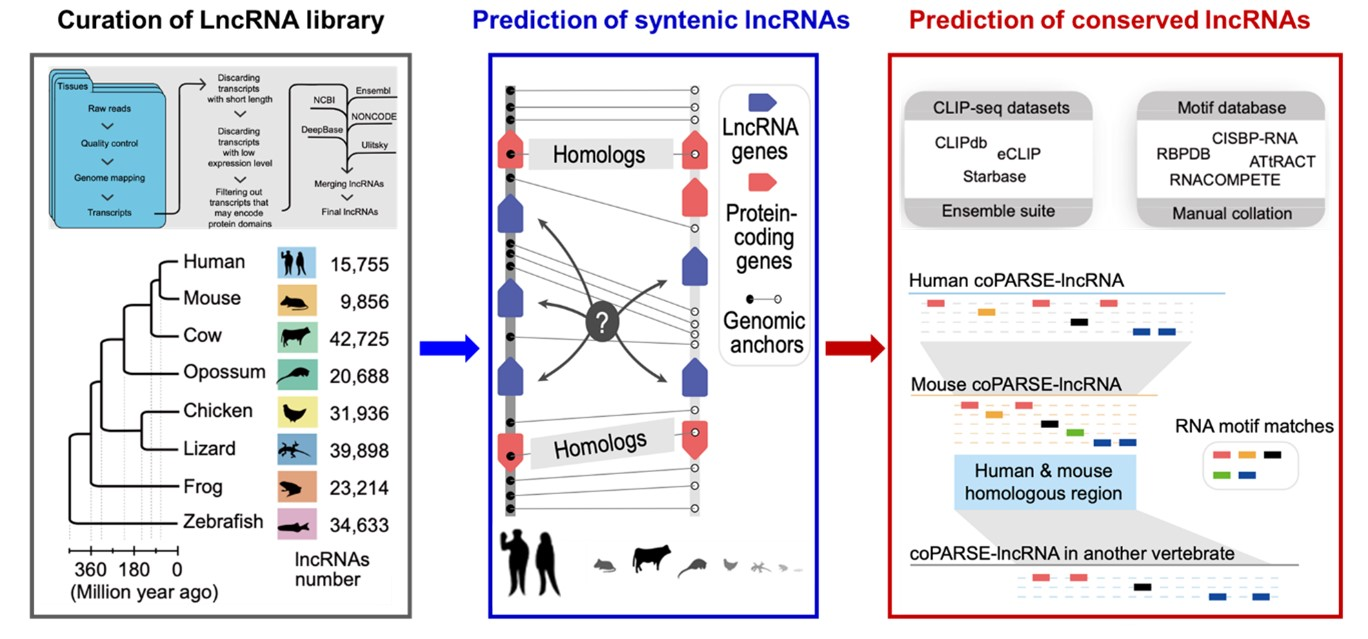
图2. 鉴定不同物种之间同源保守lncRNA方法(lncHOME)的计算流程
功能鉴定:该团队深入探究了所鉴定的同源lncRNA的功能保守性。首先,通过建立CRISPR-Cas12a介导的大片段基因敲除筛选系统,该团队鉴定出了75个能促进癌症细胞增殖的coPARSE-lncRNA,其中37个在HeLa细胞中起重要作用。随后,该团队进一步开发了一个基于CRISPR-Cas12a的敲除和回补系统,发现通过回补预测的斑马鱼同源lncRNA片段可以挽救其中4个人类coPARSE-lncRNA的敲除所导致的HeLa细胞增殖的缺陷。更有意思的是,在斑马鱼胚胎中敲低这四个斑马鱼的coPARSE-lncRNA会导致严重的胚胎发育延迟,而这些表型又可以通过回补人类的同源lncRNA进行挽救。以上结果说明这些同源lncRNA具有很强的功能保守性。
机制解析:lncHOME算法得到的同源lncRNA必然具有保守的RBP结合位点模式。根据这一条件推测,coPARSE-lncRNA具有相似的RBP结合图谱。针对其中两条coPARSE-lncRNA,该团队通过RNA沉降结合质谱实验验证了这一假设。对于上面所描述的可以挽救细胞增殖或胚胎发育缺陷的同源lncRNA片段,如果突变其中某些RBP(例如NONO和IGF2BP2)的结合位点,所得到的新的片段无法起到挽救效果。这些突变实验进一步证明了RBP结合位点对于coPARSE-lncRNA的功能的重要性。
该团队的研究提供了一套基于机器学习的计算分析方法,在脊椎动物中鉴定得到数目众多的潜在同源的lncRNA,并通过基因敲除、敲低、回补实验和结合蛋白质谱等实验验证了同源lncRNA的功能保守性。虽然这些同源lncRNA在进化过程中序列保守性逐渐消失,但是却保留着保守的RBP结合模式(图3)。该工作极大地扩展了当前脊椎动物中保守的lncRNA库,为研究lncRNA的进化、功能及作用机制的解析提供了新视角和新资源。
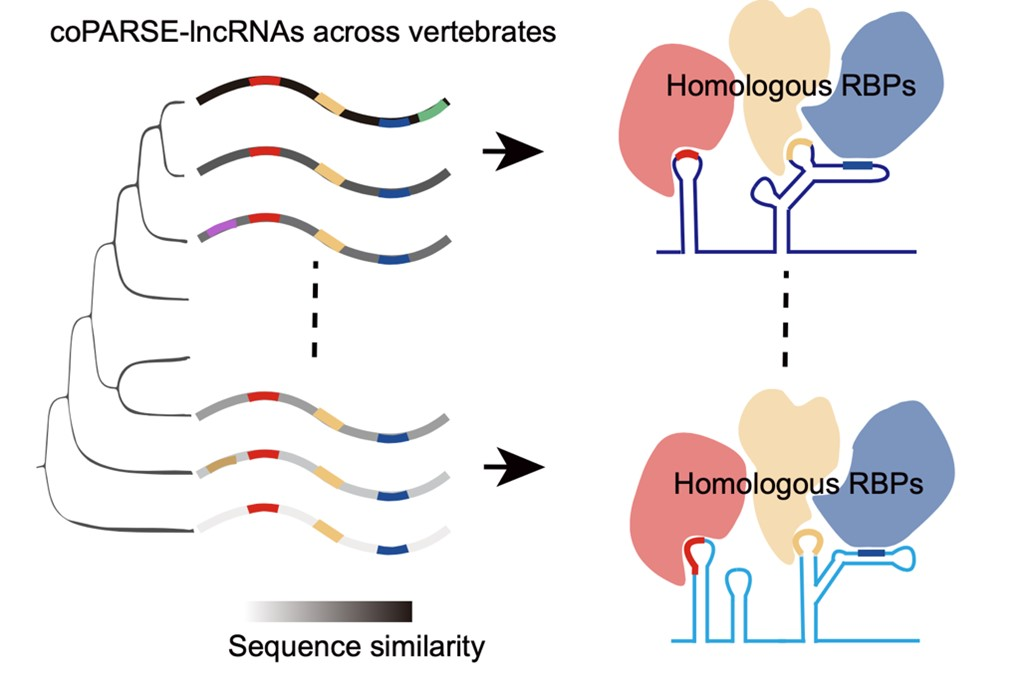
图3. 该研究鉴定的同源保守lncRNA的进化及功能机制模型
清华大学生命科学学院黄文泽博士、熊团林博士及北京大学未来技术学院赵雨亭博士为论文的共同第一作者。张强锋、汪阳明和席建忠为论文的共同通讯作者。中国科学院动物研究所刘峰研究员、衡鉴博士,清华大学生命科学学院韩鸽博士、王鹏飞博士生,北京大学赵志华博士、李娟博士、石铭和汪家震博士生、吴怡霞为论文工作作出了重要贡献。
该成果为研究lncRNA的保守性揭开了新的一页,但其重要性仍有待考验。这些资源和方法究竟能为lncRNA在其他生理病理过程中的功能提供多少借鉴仍有赖领域内科学家们的共同努力。此外,这些方法仍需要进一步完善,整合RNA结构和大模型,或者加入其他重要序列和特征(如小RNA结合位点、RNA修饰与编辑位点),有可能进一步完善lncRNA保守性的预测方法,从而提供全新的生物学洞见。分析更多物种(特别是非脊椎动物)的lncRNA数据,将会提供一个更为完整的lncRNA进化图谱,让我们一探占人类基因组绝大部分的“暗物质”的前世、今生和未来。
转载本网文章请注明出处